VAHTS Universal Plus DNA Library Prep Kit for MGI

PCR、PCR-Free文库构建;全基因组测序;全外显子或其他靶向捕获测序;宏基因组测序

· 兼容性广:轻松应对不同物种来源、不同起始量投入量和不同模板类型
· 操作简便:仅需根据目标插入片段大小调整片段化时间即可得到所需文库
· 文库产量高:优异的文库转化率、样本质量更好

VAHTS Universal Plus DNA Library Prep Kit for MGI是针对MGI高通量测序平台定向开发的DNA文库构建试剂盒。本试剂盒将DNA片段化、末端修复以及末端加dA尾合并为一步,产物无需纯化,直接进行接头连接、文库富集和分选,可将100 pg - 1 μg模板DNA转换成MGI高通量测序平台专用文库,无需对基因组进行机械打断,简化了建库流程、缩短了操作时间,适用于PCR文库的构建。本试剂盒可兼容不同来源和不同投入量的DNA,仅需根据目标插入片段大小调整片段化时间即可得到所需文库。试剂盒中提供的所有试剂都经过了严格的质量控制和功能验证,较大程度上保证了文库构建的稳定性和重复性。

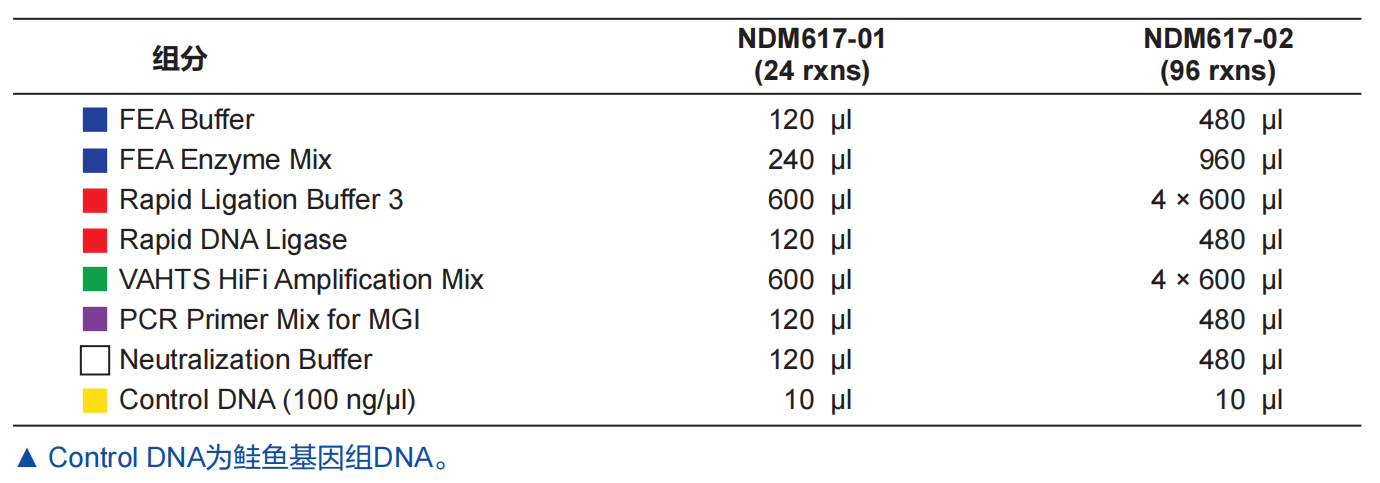

-30 ~ -15℃保存。可于-20 ~ 0℃运输。
Q1:关于Input DNA和Fragmentation
A1:a).试剂盒兼容的Input DNA量范围为50 pg - 1 μg。应尽可能使用A260/A280 = 1.8 - 2.0的高质量Input DNA。Table 1中列举了常见应用中推荐的Input DNA量。
Table 1. 常见应用中推荐Input DNA量
| Input DNA | Adapter:Input DNA 摩尔比 |
其他来源Adapter 工作浓度 |
Vazyme Adapter 预稀释倍数 |
| 500 ng - 1 μg | 10:1 - 20:1 | 510 μM | 不稀释 |
| 100 ng - 500 ng | 20:1 - 100:1 | 10 μM | 不稀释 |
| 25 ng - 100 ng | 50:1 - 200:1 | 5 μM | 1:2 |
| 5 ng - 25 ng | 40:1 - 200:1 | 1 μM | 1:10 |
| 100 pg - 5 ng | 60:1 - 3000:1 | 0.1-0.2 μM | 1:30-1:200 |
上表为使用高质量DNA时推荐的Input DNA量,当Input DNA质量较差时,应适当上调使用量。
b).推荐使用灭菌超纯水、0.1 × TE(low TE)或10 mM Tris-HCl(pH 8.0 - 8.5)溶解DNA样品;FEA Enzyme mix对EDTA敏感,请确认DNA样品中EDTA浓度,如打断末修反应体系中EDTA终浓度大于0.1 mM,请按照08-1操作说明对DNA样本进行预处理。
Q2:关于DNA Adapter
A2:a).针对MGI测序平台,Vazyme提供一套共96种Indexed Adapters。
#NM108为单端10bp Indexed Adapters,共96种。
b).DNA Adapter的质量和用量直接影响建库效率和文库的质量。Adapter投入量过高可能会导致Adapter 或Adapter Dimer残留;投入量不足又会影响连接效率进而导致文库产出降低。Table 2列举了不同Input DNA量推荐的Adapter使用量。
Table 2 100 pg - 1 μg Input DNA推荐的Adapter使用浓度
| 应用 | 样本类型 | 推荐Input DNA量 |
| 全基因组测序 | 复杂基因组 | 50 ng - 1 μg |
| 靶向捕获测序 | 复杂基因组 | 10 ng - 1 μg |
| 全基因组/靶向捕获测序 | FFPE DNA | ≥ 50 ng |
| 全基因组测序 | 微生物基因组 | 1 ng - 1 μg |
| 全基因组测序(PCR-Free文库) | 复杂/简单基因组 | ≥ 50 ng (不进行长度分选) ≥ 200 ng (进行长度分选) |
| 免疫共沉淀测序靶向测序 | ChIP DNA | ≥ 100 pg |
uInput DNA摩尔数可根据下述公式粗略计算:
Input DNA摩尔数(pmol) ≈ Input DNA质量(ng)/ [0.66 × Input DNA平均长度(bp)]
u推荐根据表中的浓度值或Vazyme Adapter的稀释倍数使用0.1 × TE预先稀释Adapter,这样可以保证建库过程中Adapter的使用体积为固定数值(5 μl),避免加样体积错误。
uAdapter的质量会直接影响Adapter与Input DNA的摩尔比,进而影响连接效率和文库产出。应选用优质的Adapter;使用0.1 × TE稀释和保存Adapter溶液;尽量避免反复冻融。
u提高Adapter的使用量可以在一定程度上提高文库产出,尤其当Input DNA ≤ 25 ng时。当需要优化建库效率时,可在上表推荐条件下额外尝试几个更高的Adapter使用量(推荐2 - 10倍范围内)。如Adapter液浓度限制无法提高使用量,可通过提高Adapter使用体积来尝试。如:默认Adapter使用体积为5 μl,
u当Input DNA为500 ng - 1 μg时可提高Vazyme Adapter使用量至10 μl以增加5 - 15%的文库产出。但是需注意提高接头浓度可能会增加文库中接头残留,造成测序数据浪费。
Q3:关于Adapter Ligation产物纯化
A3:a).Adapter Ligation产物需先去除过剩的Adapter,再进行后续文库扩增(PCR文库)或直接上机测序(PCR-Free文库)。默认纯化条件0.6 × (产物100 μl,磁珠60 μl)适用于绝大多数情况。如需获得Insert Size更长的文库,可通过适当降低磁珠使用量以减少小片段文库的含量,但这种调整只能粗略改变文库主峰的位置,如需要准确控制文库长度分布,可在此步纯化之后进行长度分选。
b).如之后进行文库长度分选,推荐洗脱体积为105 μl;否则,推荐洗脱体积为22.5 μl。
c).如数据显示纯化产物中Adapter或Adapter Dimer污染严重,可对其再进行一次磁珠纯化:使用灭菌超纯水将第一次纯化产物体积补至50 μl,加入50 μl磁珠(1 ×)进行第二次纯化。这样可以显著降Adapter或Adapter Dimer的残留水平,尤其是当构建PCR-Free文库时。有时可能还需要配合Adapter的使用量降低才能完全消除Adapter或Adapter Dimer的残留
Q4:关于磁珠的使用
A4:a). 试剂盒推荐使用VAHTSTM DNA Clean Beads (Vazyme #N411)进行磁珠纯化。
注意:如使用其他来源磁珠,纯化条件可能需要更改!
b). 磁珠操作通用注意事项:
磁珠使用量常用乘数“ × ”进行标识,表示相对于原始样品体积而言使用多少倍体积的磁珠。如样品原始体积为100 μl ,1 ×纯化时磁珠使用体积为1 × 100 μl = 100 μl;0.6 × / 0.2 ×分选时第一轮磁珠用量为0.6 × 100 μl = 60 μl第二轮磁珠用量0.2 × 100 μ= 20 μl。
磁珠使用量直接影响可纯化的DNA长度下限。乘数越高,可纯化的DNA长度下限越短;反之,则越长。例如:1 ×磁珠只能高效纯化长于250 bp的DNA,更短的DNA会在纯化过程中大量丢失;提高至1.8 ×后,150 bp的DNA也可进行高效纯化。
磁珠使用前应先平衡至室温(室温放置30 min),否则会导致得率下降、分选效果不佳。磁珠每次使用前都应充分振荡混匀或使用移液器上下吹打充分混匀。
样品与磁珠充分混匀后置于磁力架上进行分离时,请于溶液彻底澄清后再吸取上清,吸取上清时应余留2 - 3 μl。若不慎吸到磁珠,会造成得率下降、分选效果不佳甚至影响后续酶反应。此时可将磁珠混匀重新置于磁力架上再次分离即可。由于磁力架吸力不同等原因,默认分离时间有时可能需要延长,以彻底分离磁珠和液体。
磁珠漂洗应使用新鲜配制并平衡至室温的80%乙醇。漂洗过程中EP管应始终置于磁力架中,请勿扰动磁珠。产物洗脱前应将磁珠置于室温干燥。干燥不充分容易造成无水乙醇残留影响后续反应;过分干燥又会导致磁珠开裂进而降低纯化得率。通常情况下,室温干燥5 - 10 min 足以让磁珠充分干燥,请勿加热干燥(如置于37 ℃烘箱干燥)。
通常情况下,推荐使用洗脱液(10 mM Tris-HCl,pH 8.0 - 8.5)进行产物洗脱,这样更有利于产物的稳定保存。然而,当后续需对文库进行靶向捕获时,为了利于捕获前文库干燥浓缩且避免对后续捕获反应产生影响,应使用灭菌超纯水进行产物洗脱。
洗脱产物可于4℃稳定保存一周;长期保存时应置于-20℃,避免不必要的反复冻融。
Q5:关于长度分选
A5:a).如Input DNA分布范围较宽,建库过程中通常需要进行长度分选以控制最终文库长度分布范围。推荐使用双轮磁珠分选方案,也可通过切胶纯化的方式进行分选。
b).长度分选执行位置有多种选择,可置于End Preparation之前、Adapter Ligation之后或Library Amplification之后。标准实验方案中不包含长度分选步骤。如需进行,请参考附一、双轮磁珠分选进行长度分选,DNA损失量较大。有时需要在文库长度分布(进行长度分选)和文库复杂度(不进行长度分选)之间进行选择。当Input DNA量较低时,应保证长度分选执行位置的唯一性,进行两次或者两次以上的长度分选会导致文库复杂度和产出严重下降!
c).过度扩增的文库进行长度分布检测时,常见高分子量位置出现拖带或尾峰。对应产物多为非互补链交叉退火产物(参考06-6/关于Library Amplification)。推荐解决方案为调整扩增循环数,避免过度扩增,不建议通过长度分选去除拖带或尾峰。
d).Rapid Ligation Buffer 3中包含的高浓度PEG会对双轮磁珠分选和切胶纯化产生显著影响。因此,如在Adapter Ligation之后进行长度分选,Adapter Ligation产物纯化步骤(08/标准方案/步骤二/6.使VAHTS DNA Clean Beads对反应产物进行纯化)不可省略,将纯化产物洗脱至合适体积的洗脱液中,再进行双轮磁珠分选或切胶纯化;如确需在Adapter Ligation之后分选,分选条件需另行摸索。如在Library Amplification之后进行长度分选,可将原处纯化步骤直接替换为双轮磁珠分选或切胶纯化
Q6:关于Library Amplification
A6:a). PCR Primer Mix适用于扩增含完整长度Adapter的MGI高通量测序平台文库。推荐每条引物的扩增终浓度为5 - 20 μM。
b).PCR反应后期,引物通常会先于dNTP被耗尽。此时,过多的循环会造成扩增产物解链后进行非特异性退火,产生非互补链交叉退火产物。这些产物在依赖电泳的检测方式中迁移速率比较慢,在高分子量区域呈现弥散分布。它们是由正确长度的单链文库构成,在变性后能够与Flow Cell正常结合并被测序。因此,其存在与否对测序而言并无显著影响。然而,这种产物的存在对于文库定量方式的选择会产生决定性影响。因产物不是完整的双链结构,当使用基于识别双链DNA的荧光染料(Equalbit dsDNA HS Assay Kit, Vazyme #EQ111等)来进行文库定量时,定量结果会比实际值偏低;但基于qPCR的文库定量体系(如VAHTSTM Library Quantification Kit for MGI,Vazyme #NQM101 - NQ106)在定量过程中包含变性过程,仍然可以准确定量这种过度扩增的文库。
c).Library Amplification步骤需要严格控制扩增循环数。循环数不足,会导致文库产出不足;循环数过多,又会导致过度扩增、偏好性增加、重复度增加、嵌合产物增加、扩增突变积累等多种不良后果。Table 3列举了当使用50 pg - 1 μg高质量Input DNA时,获得100 ng或1 μg文库推荐的扩增循环数。
Table 3. 50 pg - 1 μg Input DNA扩增循环数推荐表
| Input DNA | Number of cycles required to generate 100 ng |
Number of cycles required to generate 1 μg |
|
50 pg 100 pg 1 ng 10 ng 50 ng 100 ng 250 ng 500 ng 1 μg |
15 - 17 14 - 16 10 - 12 5 - 7 3 - 5 1 - 3 0 0 0 |
17 - 19 15 - 17 12 - 14 8 - 10 5 - 7 4 - 6 3 - 5 3 - 4 1 - 3 |
上表为使用高质量的小鼠gDNA 37℃ 片段化8 min建库测得的循环数参数。当DNA质量较差时,
需适当调整循环数以获取足量文库。
若连接接头后进行长度分选,则参照较高循环数进行Library Amplification;否则,参照较低循环
数即可。
Q7:关于文库的质量控制
通常情况下,构建好的文库可通过长度分布检测和浓度检测来进行质量评价。
A7:a). 文库长度分布检测:
文库长度分布可通过LabChip® GX、GXII、GX Touch (PerkinElmer);Bioanalyzer、Tapestation (Agilent Technologies);Fragment Analyzer™ (Advanced Analytical)等基于电泳分离原理的设备进行检测。
常规适用于MGI平台的Adapter均为“Ω”型Adapter,部分区域存在单链分叉区域。因此,Adapter Ligation之后未经扩增的PCR-Free文库两端都存在单链分叉区域。当进行文库长度分布检测时,这种单链的分叉结构会造成文库迁移速率变慢,进而导致检测结果偏长、分布范围变宽、峰型异常等问题。为了准确检测文库长度分布,可取少量PCR-Free文库进行适度PCR扩增,取扩增产物进行检测以反映PCR-Free文库的长度分布情况。也可通过检测VAHTS®Library Quantification Kit for MGI (Vazyme #NQM101 - NQM106)扩增产物来反映PCR-Free文库的长度分布情况
b). 文库浓度检测:
常用文库浓度检测方法有两种:基于双链DNA荧光染料的方法,如Equalbit dsDNA HS Assay Kit (Vazyme #EQ111)、Equalbit® 1 × dsDNA HS Assay Kit(Vazyme #EQ121)、Qubit®、PicoGreen®等;基于qPCR绝对定量的方法,如VAHTS®Library Quantification Kit for MGI (Vazyme #NQM101 - NQM106)。虽然前者简单易行,但由于下述原因,推荐使用基于qPCR绝对定量的方法进行文库浓度测定。
当使用完整长度Adapter,且完成Adapter Ligation步骤之后,任何一个步骤产物都可以使用基于qPCR绝对定量的方法进行浓度测定。可以明确监控接头连接、磁珠纯化/分选、Library Amplification各步骤效率,进而能为体系优化和建库异常原因分析提供依据.
因PCR-Free文库缺少Library Amplification步骤,构建好的文库中包含一定比例的单端连接Adapter的产物和两端都未连接Adapter的产物。当使用Qubit®、PicoGreen®等基于双链DNA荧光染料的浓度测定方法时,无法有效区分这些产物。但qPCR绝对定量基于PCR扩增原理,只定量样品中两端Adapter完整的文库(即可测序文库),可以排除单端或双端都未连接Adapter的不可测序文库的干扰。因此,PCR-Free文库只能使用基于qPCR绝对定量的方法进行浓度测定。
过度扩增的文库因包含大量不完整双链结构,不能使用Qubit®、PicoGreen®等基于双链DNA荧光染料的浓度测定方法。但基于qPCR绝对定量的方法不受过度扩增影响(参考06-6/关于Library Amplification)
Q8.其他注意事项
A8:a).DNA片段化产物的大小和分布范围是由时间依赖的酶促反应决定的,因此配制片段化反应体系时应在冰上操作。
b).使用前请将试剂盒各组分置于室温解冻。解冻后上下颠倒数次充分混匀,短暂离心后置于冰上待用。为避免样品交叉污染,推荐使用带滤芯的枪头,吸取不同样品时请更换枪头。
c).推荐在带热盖的PCR仪中进行各步骤反应,使用前应预热PCR仪至反应温度附近。
d).PCR产物因操作不当极容易产生气溶胶污染,进而影响实验结果准确性。因此,我们推荐将PCR反应体系配制区和PCR产物纯化检测区进行强制性的物理隔离;使用专用的移液器等设备;并定时对各实验区域进行清洁(使用0.5%次氯酸钠或10%漂白剂进行擦拭清理),以保证实验环境的洁净度。
相关产品
VAHTS DNA Adapters Set 8 for MGI
NM108-01/02
N411-01/02/03
EQ111-01/02
Equalbit 1 × dsDNA HS Assay Kit
EQ121-01/02





